溶菌微生物 Flexibacter sp.FL824Aのキチン分解酵素遺伝子の解析
Flexibacter sp. FL824A の染色体DNAから、2つの活性ドメインを持つ特異な構造のキチナーゼの遺伝子と、エンド-β-N-アセチルグルコサミニダーゼの遺伝子の2つのキチン分解酵素遺伝子をクローニングし、全塩基配列を決定した。
背景・ねらい
牧草病害において生物機能を利用した病害防除技術の開発研究はほとんど進んでいないことから、牧草での微生物間の相互作用を解明し、それを利用した病害の防除技術の開発が世界的に望まれている。これまでに、作物地上部から植物病原糸状菌を強く溶菌する細菌Flexibacter sp. FL824A を分離している。しかし、その溶菌機構については不明でのままである。そこで、溶菌機構の中で重要な役割を担っていると考えれられるキチン分解酵素の遺伝子を明らかにし、その成果を溶菌生物を利用した病害防除技術の開発に資することを目的とする。
成果の内容・特徴
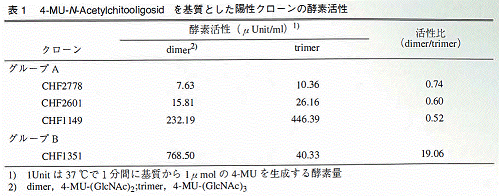
- Flexibacter sp.の染色体DNAからpUC19と大腸菌を用い、4MU-(G1cNAc)2と4MU-(G1cNAc)3を基質として粗タンパク抽出液の分解活性を測定することにより、4株の陽性クローンを得た。それらは、基質特異性により2つのグループに分類できた(表1)。活性の大きさ、基質特異性、インサートの大きさおよび制限酵素による切断パターンからクローンCHF1149とCHF1351を選び出し、各々の挿入断片の制限酵素地図を作成して、少なくとも異なる2種類のキチン分解遺伝子がクローニングされたと結論した。
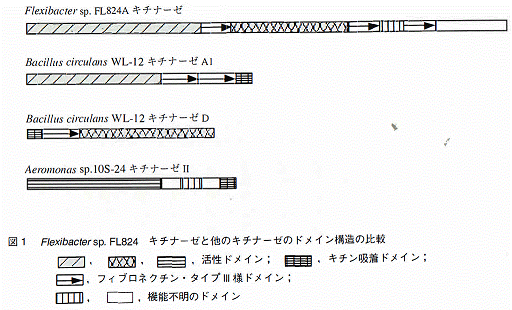
- クローンCHF1149の挿入断片の全長8.7kbの全塩基配列を決定し、1412アミノ酸からなるタンパクを推定した。本タンパクは、N末端側に Bacillus circulans WL-12のキチナーゼA1の活性ドメインと、C末端側に同菌のキチナーゼDのそれと相同性を有する2つの活性ドメインを持つ特異な構造のキチナーゼであることがわかった(図1)。
- クローンCHF1351については、サブクローニング後、全長5.0kbのDNA断片の全塩基配列を決定し、654アミノ酸からなるタンパク質を推定した。これは、Clostridium perfringensα のエンド-β-N-アセチルグルコサミニダーゼと相同性(25%、アミノ酸レベル)があった。
成果の活用面・留意点
Flexibacter sp. FL824Aの強い溶菌能力は、一つとして2つの活性ドメインをもつキチナーゼによると考えられるが、本キチナーゼの分解特性は不明のままであるので、その特性を明らかにする必要がある。
具体的データ
- Affiliation
-
国際農研 畜産草地部
- 分類
-
研究
- 予算区分
- 大型別枠(生態秩序)
- 研究課題
-
植物病害菌に対する溶菌微生物の拮抗機構の解明と利用技術の開発
- 研究期間
-
平成8~10年
- 研究担当者
-
安藤 康雄 ( 畜産草地部 )
- ほか
- 発表論文等
-
取りまとめ中。
- 日本語PDF
-
1998_11_A3_ja.pdf417.38 KB